数据挖掘实验五
一、实验目的
- 掌握聚类算法: $K-means$。
- 掌握聚类算法: $AGNES$。
- 掌握聚类算法: $DBSCAN$。
二、实验要求
通过本实验应达到如下的要求:
- 掌握三种聚类算法的基本思想
- 熟练使用 $Python$ 或其他工具来实现聚类算法。
三、实验器材
- 计算机一台。
- $Python$ 或其他变成工具。
四、实验内容
数据集的选取
分散性聚类(Kmeans)
算法思想
$K-Means$ 算法的思想很简单,对于给定的样本集,按照样本之间的距离大小,将样本集划分为 $K$ 个簇。让簇内的点尽量紧密的连在一起,而让簇间的距离尽量的大(即是把一堆一堆给分开)。
如果用数据表达式表示,假设簇划分为$(C_1,C_2,…,C_k)$,则我们的目标是最小化平方误差E: \(E = \sum_{i=1}^{k}\sum_{x∈C_i}||x\ -\ u_i||^2\) 其中 $u_i$ 是簇 $C_i$ 的均值向量,有时也被称为质心,表达式为: \(u_i\ =\ \frac{1}{|C_i|}\sum_{x∈C_i}x\) 如果我们想直接求上式的最小值并不容易,这是一个 $NP$ 难度的问题,因此只能采用启发式的迭代方法。
$K-Means$ 采用的启发式方式很简单,用下面一组图就可以形象的描述。
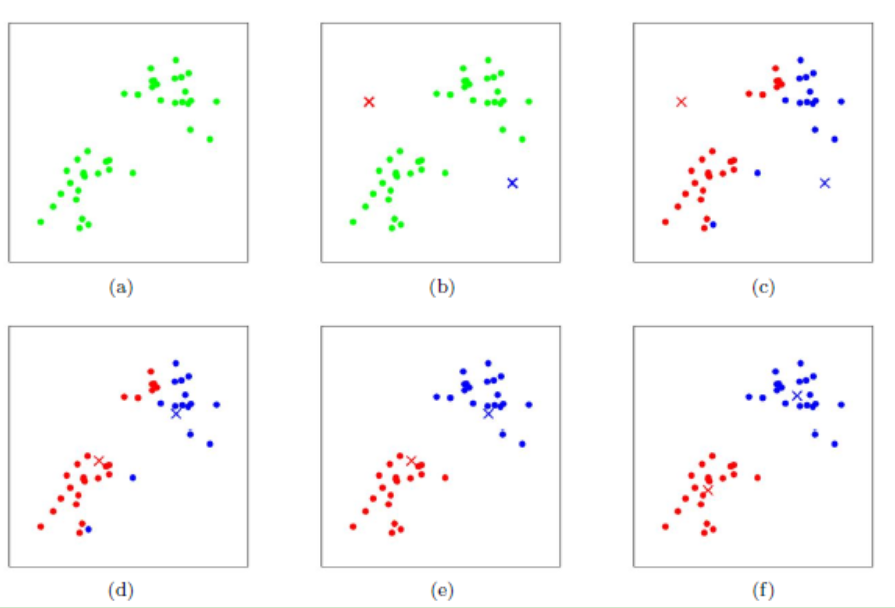
上图 $a$ 表达了初始的数据集,假设 $k=2$。在图 $b$ 中,我们随机选择了两个 $k$ 类所对应的类别质心,即图中的红色质心和蓝色质心,然后分别求样本中所有点到这两个质心的距离,并标记每个样本的类别为和该样本距离最小的质心的类别,如图 $c$ 所示,经过计算样本和红色质心和蓝色质心的距离,我们得到了所有样本点的第一轮迭代后的类别。此时我们对我们当前标记为红色和蓝色的点分别求其新的质心,如图 $d$ 所示,新的红色质心和蓝色质心的位置已经发生了变动。图 $e$ 和图 $f$ 重复了我们在图c和图d的过程,即将所有点的类别标记为距离最近的质心的类别并求新的质心。最终我们得到的两个类别如图f。
当然在实际 $K-Mean$ 算法中,我们一般会多次运行图 $c$ 和图 $d$ ,才能达到最终的比较优的类别。
算法流程
对于 $K-Means$ 算法,首先要注意的是 $K$ 值的选择,一般来说,我们会根据对数据的先验经验来选择一个合适的 $K$ 值,如果没有什么先验知识,则可以通过交叉验证选择一个合适的 $K$ 值。
在确定了 $K$ 的个数后,我们需要选择 $K$ 个初始化的质心,就像上图 $b$ 中的随机质心。由于我们是启发式方法,$K$ 个初始化的质心的位置选择对最后的聚类结果和运行时间都有很大的影响,因此需要选择合适的 K 个质心,最好这些质心不能靠的太近。
算法流程如下:
输入是样本集$D={x_1,x_2,…x_m}$ 聚类的簇树 $K$ ,最大迭代次数 $N$。
输出是簇划分 $C\ =\ {C_1,C_2,……C_k}$。
-
从数据集 D 中随机选择 K 个样本作为初始的 K 个质心向量:${μ_1,μ_2, …, u_k}$。
-
对于 $n=1,2, …,N$。
a) 将簇划分 C 初始化为 $C_t = ∅$。
b) 对于$i=1,2…m$,计算样本$x_i$和各个质心向量$μ_j(j=1,2,…k)$ 的距离:$d_{ij}= x_i−μ_j ^2$,将$x_i$标记最小的为 $d_{ij}$ 所对应的类别$λ_i$。此时更新 $C_{λi}=C_{λ_i}∪{x_i}$。 c) 对于$j=1,2,…,k$,对$C_j$中所有的样本点重新计算新的质心
e) 如果所有的k个质心向量都没有发生变化,则转到步骤3)。
3.输出簇划分$C={C1,C2,…Ck}$。
优点
- 原理比较简单,实现容易,收敛速度快。
- 聚类效果较优。
- 算法的可解释度比较强。
- 主要需要调参的参数仅仅是簇数 $K$。
缺点
- $K$ 值得选取不好把握。
- 对于不是凸得数据集比较难收敛。
- 如果各隐含类别的数据不平衡,比如各隐含类别的数据量严重失衡,或者各隐含类别的方差不同,则聚类效果不佳。
- 采用迭代方法,得到的结果只是局部最优。
- 对噪音和异常点比较的敏感。
K-Means++
在上节我们提到,k个初始化的质心的位置选择对最后的聚类结果和运行时间都有很大的影响,因此需要选择合适的k个质心。如果仅仅是完全随机的选择,有可能导致算法收敛很慢。K-Means++算法就是对K-Means随机初始化质心的方法的优化。
K-Means++的对于初始化质心的优化策略也很简单,如下:
- 从输入的数据点集合中随机选择一个点作为第一个聚类中心 $μ1$。
-
对于数据集中的每一个点 $x_i$,计算它与已选择的聚类中心中最近聚类中心的距离$D(x_i)=argmin( xi−μr )^22\ r=1,2,…k{selected}$。 - 选择一个新的数据点作为新的聚类中心,选择的原则是:$D(x)$ 较大的点,被选取作为聚类中心的概率较大。
- 重复 $2$ 和 $3$ 直到选择出k个聚类质心。
- 利用这 $K$ 个质心来作为初始化质心去运行标准的 $K-Means$ 算法。
大样本优化 Mini Batch K-Means
在统的 $K-Means$ 算法中,要计算所有的样本点到所有的质心的距离。如果样本量非常大,比如达到 $10$ 万以上,特征有 $100$ 以上,此时用传统的 $K-Means$ 算法非常的耗时,就算加上 $elkan K-Means$ 优化也依旧。在大数据时代,这样的场景越来越多。此时 $Mini\ Batch\ K-Means$ 应运而生。
顾名思义,$Mini Batch$,也就是用样本集中的一部分的样本来做传统的 $K-Means$,这样可以避免样本量太大时的计算难题,算法收敛速度大大加快。当然此时的代价就是我们的聚类的精确度也会有一些降低。一般来说这个降低的幅度在可以接受的范围之内。
在 $Mini\ Batch\ K-Means$中,我们会选择一个合适的批样本大小 $batch\ size$,我们仅仅用 $batch\ size$ 个样本来做 $K-Means$ 聚类。那么这 $batch\ size$ 个样本怎么来的?一般是通过无放回的随机采样得到的。
为了增加算法的准确性,我们一般会多跑几次 $Mini\ Batch\ K-Means$ 算法,用得到不同的随机采样集来得到聚类簇,选择其中最优的聚类簇。
K-Means与KNN
初学者很容易把 $K-Means$ 和 $KNN$ 搞混,两者其实差别还是很大的。
$K-Means$ 是无监督学习的聚类算法,没有样本输出;而 $KNN$ 是监督学习的分类算法,有对应的类别输出。$KNN$ 基本不需要训练,对测试集里面的点,只需要找到在训练集中最近的 $k$ 个点,用这最近的 $k$ 个点的类别来决定测试点的类别。而 $K-Means$ 则有明显的训练过程,找到 $k$ 个类别的最佳质心,从而决定样本的簇类别。
当然,两者也有一些相似点,两个算法都包含一个过程,即找出和某一个点最近的点。两者都利用了最近邻 $(nearest neighbors)$ 的思想。
聚类个数
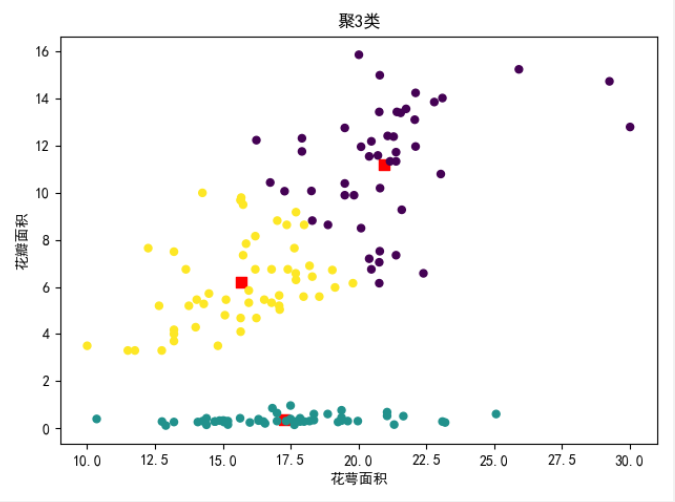
在本次实验中, 取 $K =3$。
代码
数据的获取。
def shuju(k):
b = set()
while (len(b) < k):
b.add(np.random.randint(0, 150))
return (b)
计算点到中心点的距离,返回一个列表。
def getDistance(point_x, point_y, cent_x, cent_y, k):
x = point_x
y = point_y
x0 = cent_x
y0 = cent_y
i = 0
j = 0
ds = [[] for i in range(len(x))]
while i < len(x):
while j < k:
M = np.sqrt((x[i] - x0[j]) * (x[i] - x0[j]) + (y[i] - y0[j]) * (y[i] - y0[j]))
M = round(M, 1)
j = j + 1
ds[i].append(M)
j = 0
i = i + 1
return (ds)
计算点到中心点的误差。
def EDistance(point_x, point_y, cent_x, cent_y, k):
x = point_x
y = point_y
x0 = cent_x
y0 = cent_y
i = 0
j = 0
sum = 0
while i < k:
while j < len(x):
M = (x[j] - x0[i]) * (x[j] - x0[i]) + (y[j] - y0[i]) * (y[j] - y0[i])
M = round(M, 1)
sum += M
j = j + 1
# ds[i].append(M)
j = 0
i = i + 1
return (sum)
确定中心点。
def cent(lable): #获取一组数据的中心点
temp = lable
mean_x = []
mean_y = []
i = 0
j = 0
while i < 3:
cent_x = 0
cent_y = 0
count = 0
while j < len(x):
if i == temp[j]:
count = count + 1
cent_x = cent_x + x[j]
cent_y = cent_y + y[j]
j = j + 1
cent_x = cent_x / count
cent_y = cent_y / count
# 更新中心点
mean_x.append(cent_x)
mean_y.append(cent_y)
j = 0
i = i + 1
return [mean_x, mean_y]
按照 $K$ 值的大小进行聚类,其中 $ds$ 指的是存有许多点到中心点的距离的列表。
# 按照k值聚类
def julei(ds, x):
x = x
x = len(x)
i = 0
temp = []
while i < x:
temp.append(ds[i].index(min(ds[i])))
i = i + 1
return (temp)
#ds[0][0] 指的是index 为 0 的点到第一个中心点的距离
当中心点的位置不再发生变化时跳出。
while np.abs(temp2 - temp1) != 0: #中心不再发生调整时跳出
temp1 = temp2
center = cent(temp)
x0 = center[0]
y0 = center[1]
ds = getDistance(x, y, x0, y0, k)
temp = julei(ds, x)
temp2 = EDistance(x, y, x0, y0, k)
n = n + 1
print(n, temp2)
可视化图
层次聚类(AGNES)
AGNES 算法是一种基于层次凝聚的聚类算法,它的思想十分的朴素。假设现在有一个待聚类的数据集,那么根据分而治之的思想,我们可以首先将每一个样本点看成是一个类,然后根据一定的规则将这些比较“小”的类进行合并,进而达到最终想要的结果。
那么这个合并的规则是什么呢?通常我们将样本点之间的距离看成相似度。在两个小类中,第一个类和第二个类中的点它们之间的距离有很多,如果第一个类有 n 个样本点,第二个类有 m 个样本点,那么不同的类点和点之间的距离就会有 m*n 个,那么如何定义这个规则呢?
一般来说有三种方式可以采用,即最小距离,最大距离,和平均距离。最小距离就是分处在两个小类中的距离最近的两个点,最大距离就是指分出在两个小类中的距离最远的,平均距离就是将两个类之间的点的所有距离加权求和,即所有距离除以距离的个数。
分类
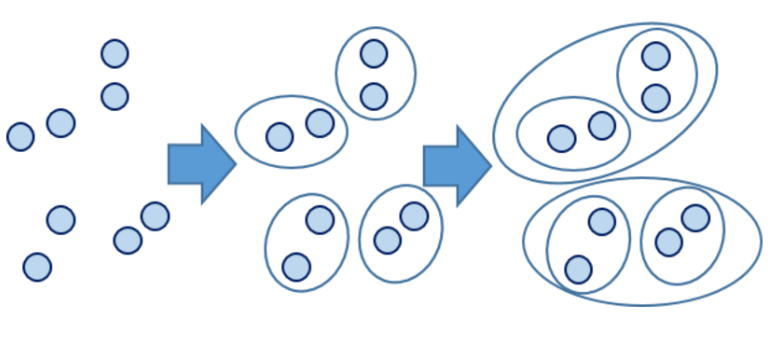
凝聚的层次聚类:一种自底向上的策略,首先将每个对象作为一个簇,然后合并这些原子簇为越来越大的簇,直到某个终结条件被满足。
分裂的层次聚类:采用自顶向下的策略,它首先将所有对象置于一个簇中,然后逐渐细分为越来越小的簇,直到达到了某个终结条件。
其中,层次凝聚的代表是 $AGNES(AGglomerative\ NESting)$ 算法。$AGNES$ 算法最初将每个对象作为一个簇,然后这些簇根据某些准则被一步步地合并。两个簇间的相似度有多种不同的计算方法。聚类的合并过程反复进行直到所有的对象最终满足簇数目。
算法步骤
- 输入:包含 $n$ 个对象的数据库。
- 输出:满足终止条件的若干个簇。
- 将每个对象当作一个初始簇。
- 重复下序操作。
- 计算任意两个簇的距离,并找到最近的两个簇。
- 合并两个簇,生成新的簇的集合。
- 达到终止条件时满足。
距离计算
$Single Linkage$
$Single Linkage$ 的计算方法是将两个组合数据点中距离最近的两个数据点间的距离作为这两个组合数据点的距离。这种方法容易受到极端值的影响。两个很相似的组合数据点可能由于其中的某个极端的数据点距离较近而组合在一起。
$Complete Linkage$
$Complete Linkage$ 的计算方法与 $Single Linkage $相反,将两个组合数据点中距离最远的两个数据点间的距离作为这两个组合数据点的距离。
$Complete Linkage$ 的问题与 $Single Linkage$ 相同,两个不相似的组合数据点可能由于 其中的极端值距离过远而永远无法组合在一起。
$Average Linkage$
$Average Linkage$ 的计算方法是计算两个组合数据点中的每个数据点与其他所有数据点的距离。将所有距离的均值作为两个组合数据点间的距离。这种方法计算量比较大,但结果比前两种方法更合理。
我们使用 $Average Linkage$ 计算组合数据点间的距离。下面是计算组合数据点 $(A,F)$ 到 $(B,C)$ 的距离,这里分别计算了 $(A,F)$ 和 $(B,C)$ 两两间距离的均值。 \(D = \frac{\sqrt{(A-B)^{2}}+\sqrt{(A-C)^{2}}+\sqrt{(F-B)^{2}}+\sqrt{(F-C)^{2}}}{4}\)

终止条件
- 设定一个最小距离阈值 $\hat{d}$,如果最相近的两个簇的距离已经超过了 $\hat{d}$,则它们不再需要合并,聚类终止。
- 限定簇的个数 $k$,当得到的簇的个数已经达到 $k$,则聚类终止。
优点
- 距离和规则的相似度容易定义少,限制少。
- 不需要预先指定聚类数。
- 可以发现类的层次关系。
缺点
- 计算复杂度太高。
- 奇异值也能产生很大影响。
- 算法很可能聚类成链状。
代码
通过双重循环找到距离最小的两个类。
def findmin(mat): # 找出距离最小的两个类
temp = mat[0][1]
x = 0
y = 0
for i in range(mat.shape[0]): #双重循环找最小的两个类,
for j in range(mat.shape[1]):
if mat[i][j] < temp and i != j:
x = i
y = j
temp = mat[i][j]
return [x, y, temp]
计算距离时,采用最大距离。
def dis(X, a, b): # 采用最大距离
temp = 0
for i in a:
for j in b:
T = np.linalg.norm(X[i] - X[j], ord=2)
if temp < T:
temp = T
return temp
AGNES 算法
def AGNES(X, k):
C = []
m = len(X)
for j in range(m):
C.append([j])
M = np.zeros((m, m)) #创造 m x m 的 0 矩阵
for i in range(m):
for j in range(i):
M[i][j] = np.linalg.norm(X[i] - X[j], ord=2) #i 到 j 的距离
M[j][i] = M[i][j]
q = m
while q > k:
[x, y, m] = findmin(M)
C[x].extend(C[y])
C.pop(y)
M = np.delete(M, y, axis=0) # 删除第J行和第J列
M = np.delete(M, y, axis=1)
for j in range(q - 1):
M[x][j] = dis(X, C[x], C[j]) # 更新距离(这一步可以优化,因为在代码开始阶段就已将所有点之间的距离计算完成。)
M[j][x] = M[x][j]
q = q - 1
return C
简单的进行一个图像绘制,并无特别。
def plot(X, c):
for i in c:
x = []
y = []
for j in i:
x.append(X[j][0])
y.append(X[j][1])
plt.scatter(x, y)
plt.show()
可视化图
密度聚类(DBSCAN)
算法原理
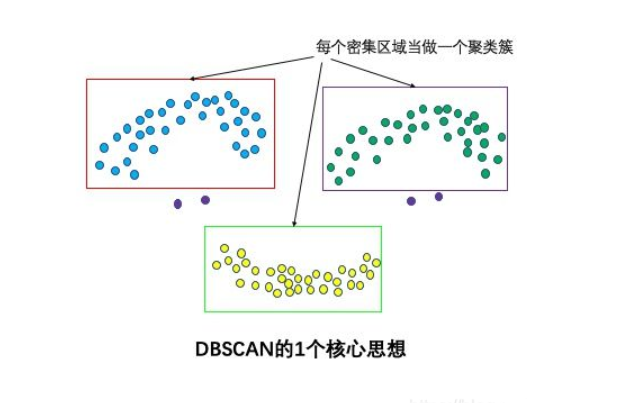
$DBSCAN$ 是一种基于密度的聚类算法,这类密度聚类算法一般假定类别可以通过样本分布的紧密程度决定。
同一类别的样本,它们之间是紧密相连的。也就是说,在该类别任意样本的周围不远处一定有同类别的样本存在。
通过将紧密相连的样本化为一类,这样就得到了一个聚类类别。通过将所有各组紧密相连的样本划为各个不同的类别,则我们就得到了最终的所有聚类类别结果。
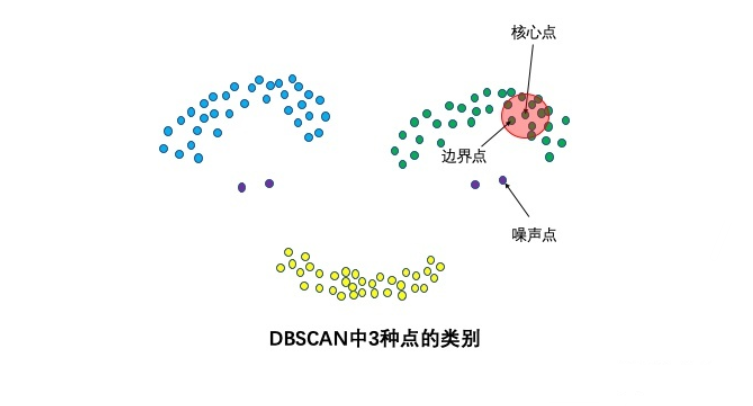
点的类别
邻域半径R内样本点的数量大于等于minpoints的点叫做核心点。不属于核心点但在某个核心点的邻域内的点叫做边界点。既不是核心点也不是边界点的是噪声点。
密度定义
$DBSCAN$ 是基于一组邻域来描述样本集的紧密程度的,参数 $(ϵ, MinPts)$ 用来描述邻域的样本分布紧密程度。其中,$ϵ$ 描述了某一样本的邻域距离阈值,$MinPts$ 描述了某一样本的距离为ϵ的邻域中样本个数的阈值。
假设我的样本集是 $D=(x_1,x_2,…,x_m)$ ,则 $DBSCAN$ 具体的密度描述定义如下:
-
$ϵ-$ 邻域:对于$x_j∈D$ ,其 $ϵ-$ 邻域包含样本集 $D$ 中与 $x_j$ 的距离不大于 $ϵ$ 的子样本集,即$N_ϵ(x_j)={x_i∈D distance(x_i,x_j)≤ϵ}$, 这个子样本集的个数记为$ N_ϵ(x_j) $。 -
核心对象:对于任一样本 $x_j∈D$,如果其 $ϵ-$ 邻域对应的 $N_ϵ(x_j)$ 至少包含 $MinPts$ 个样本,即如果 $ N_ϵ(x_j) ≥MinPts$,则 $x_j$ 是核心对象。 - 密度直达:如果 $x_i$ 位于 $x_j$ 的 $ϵ-$ 邻域中,且 $x_j$ 是核心对象,则称 $x_i$ 由 $x_j$ 密度直达。注意反之不一定成立,即此时不能说 $x_j$ 由 $x_i$ 密度直达, 除非且 $x_i$ 也是核心对象。
- 密度可达:对于 $x_i$ 和 $x_j$,如果存在样本样本序列 $p_1,p_2,…,p_T$,满足 $p_1=x_i,p_T=x_j$, 且 $p_{t+1}$ 由 $p_t$ 密度直达,则称 $x_j$ 由 $x_i$ 密度可达。也就是说,密度可达满足传递性。此时序列中的传递样本 $p_1,p_2,…,p_{T−1}$均为核心对象,因为只有核心对象才能使其他样本密度直达。注意密度可达也不满足对称性,这个可以由密度直达的不对称性得出。
- 密度相连:对于 $x_i$ 和 $x_j$ ,如果存在核心对象样本 $x_k$,使 $x_i$和 $x_j$ 均由 $x_k$ 密度可达,则称 $x_i$ 和 $x_j$ 密度相连。注意密度相连关系是满足对称性的。
算法思想
$DBSCAN$ 的聚类定义很简单:由密度可达关系导出的最大密度相连的样本集合,即为我们最终聚类的一个类别,或者说一个簇。
这个 $DBSCAN$ 的簇里面可以有一个或者多个核心对象。如果只有一个核心对象,则簇里其他的非核心对象样本都在这个核心对象的 $ϵ-$ 邻域里;如果有多个核心对象,则簇里的任意一个核心对象的 $ϵ-$ 邻域中一定有一个其他的核心对象,否则这两个核心对象无法密度可达。这些核心对象的ϵ-邻域里所有的样本的集合组成的一个$DBSCAN$ 聚类簇。
那么怎么才能找到这样的簇样本集合呢?$DBSCAN$ 使用的方法很简单,它任意选择一个没有类别的核心对象作为种子,然后找到所有这个核心对象能够密度可达的样本集合,即为一个聚类簇。接着继续选择另一个没有类别的核心对象去寻找密度可达的样本集合,这样就得到另一个聚类簇。一直运行到所有核心对象都有类别为止。
基本上这就是 $DBSCAN$ 算法的主要内容了,是不是很简单?但是我们还是有三个问题没有考虑。
第一个是一些异常样本点或者说少量游离于簇外的样本点,这些点不在任何一个核心对象在周围,在$DBSCAN$ 中,我们一般将这些样本点标记为噪音点。
第二个是距离的度量问题,即如何计算某样本和核心对象样本的距离。在 $DBSCAN$ 中,一般采用最近邻思想,采用某一种距离度量来衡量样本距离,比如欧式距离。这和 $KNN$ 分类算法的最近邻思想完全相同。对应少量的样本,寻找最近邻可以直接去计算所有样本的距离,如果样本量较大,则一般采用 $KD$ 树或者球树来快速的搜索最近邻。
第三种问题比较特殊,某些样本可能到两个核心对象的距离都小于 $ϵ$ ,但是这两个核心对象由于不是密度直达,又不属于同一个聚类簇,那么如果界定这个样本的类别呢?一般来说,此时 $DBSCAN$ 采用先来后到,先进行聚类的类别簇会标记这个样本为它的类别。也就是说 $DBSCAN$ 的算法不是完全稳定的算法。
算法流程
- 输入:样本集 $D=(x_1,x_2,…,x_m)$,邻域参数$(ϵ,MinPts)$, 样本距离度量方式。
- 输出: 簇划分 $C$ 。
- 初始化核心对象集合 $Ω=∅$ , 初始化聚类簇数 $k=0$ ,初始化未访问样本集合 $Γ = D$ , 簇划分 $C = ∅$
- 对于 $j=1,2,…m$, 按下面的步骤找出所有的核心对象:
- 通过距离度量方式,找到样本 $x_j$ 的 $ϵ-$ 邻域子样本集 $N_ϵ(x_j)$。
-
如果子样本集样本个数满足 $ N_ϵ(x_j) ≥MinPts$,将样本 $x_j$ 加入核心对象样本集合:$Ω=Ω∪{x_j}$。
- 如果核心对象集合 $Ω=∅$ ,则算法结束,否则转入步骤4.
- 在核心对象集合 $Ω$ 中,随机选择一个核心对象 $o$,初始化当前簇核心对象队列 $Ω_{cur}={o}$, 初始化类别序号$k=k+1$ ,初始化当前簇样本集合 $C_k={o}$, 更新未访问样本集合 $Γ=Γ−{o}$。
- 如果当前簇核心对象队列 $Ω_{cur}=∅$ ,则当前聚类簇 $C_k$ 生成完毕, 更新簇划分 $C={C_1,C_2,…,C_k}$ , 更新核心对象集合 $Ω=Ω−C_k$, 转入步骤3。否则更新核心对象集合 $Ω=Ω−C_k$。
- 在当前簇核心对象队列 $Ω_{cur}$ 中取出一个核心对象 $o′$,通过邻域距离阈值 $ϵ$ 找出所有的 $ϵ-$ 邻域子样本集 $N_ϵ(o′)$ ,令 $Δ=N_ϵ(o′)∩Γ$ , 更新当前簇样本集合 $C_k=C_k∪Δ$, 更新未访问样本集合 $Γ=Γ−Δ$ , 更新 $Ω_{cur}=Ω_{cur}∪(Δ∩Ω)−o′$,转入步骤
- 输出结果为: 簇划分 $C={C_1,C_2,…,C_k}$。
优点
- 自适应的聚类,不需要提前设定 $K$ 值大小,可以自适应的做聚类结果。
- 对噪声不敏感。这时因为该算法能够较好地判断离群点,并且即使错判离群点,对最终的聚类结果也没什么影响。
- 能发现任意形状的簇。这是因为 $DBSCAN$ 是靠不断链接领域的高密度点来发现簇的,只需要定义领域大小和密度阈值,因此可以发现不同形状、不同大小的簇。
- 聚类结果没有偏倚,相对的,$K-Means$ 之类的聚类算法初始值对聚类结果有很大影响。
缺点
- 对两个参数的设置敏感,即圈的半径 $eps$、阈值 $MinPts$。
- $DBSCAN$ 使用固定的参数识别聚类。显然,当聚类的稀疏程度不同,聚类效果也有很大的不同。即数据密度不均匀时,很难使用该算法。
- 如果数据样本集越大,则收敛时间越长。此时可以使用 $KD$ 树进行优化。
代码
计算各个点之间的距离,通过矩阵存储
def distance(X): #计算个点之间的距离(矩阵,同pdist2)
n=len(X)
D=np.zeros((n,n))
for i in range(n):
for j in range(0,i):
D[i][j]=np.linalg.norm(X[i]-X[j],ord=2)
D[j][i]=D[i][j]
return D
列出点 $i $ 附近的 $ε$ 范围内的点。
def RegionQuery(i): #列出一个点周围ε内存在的点的下标
Neighbors=np.where(D[i,:]<=epsilon)
return list(Neighbors)[0].tolist() #以列表形式返回
赋予类别,并将点进行归类。
def ExpandCluster(i,Neighbors,C): #将邻域点能够成为核心点的点归类
idx[i]=C #将点赋给类别
k=1
while k<=len(Neighbors):
j=Neighbors[k]
if visited[j]==0:
visited[j]=1
Neighbors2=RegionQuery(j)
if len(Neighbors2)>=MinPts:
Neighbors.extend(Neighbors2)
if idx[j]==0:
idx[j]=C #将没有分类的点分类
k=k+1
if k>len(Neighbors)-1:
break
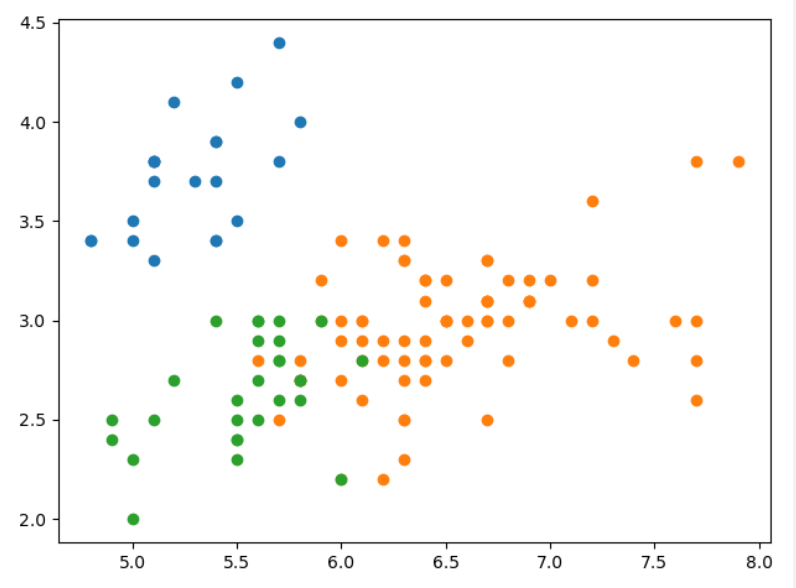
$DBSCAN$ 算法。其中 $epsilon$ ,$MinPts$ 分别设置为 $0.2$,$3$ 。
def dbscan(X,epsilon,MinPts): #领域参数分别设置为0.2 3
C=0 #以点在源数据中的位置标记类别
n=len(X)
idx=[0 for i in range(n)] #类别信息存储
visited=[0 for i in range(n)] #巡查判断
D=distance(X) #距离矩阵
for i in range(n): #寻找核心点
if visited[i]==0:
visited[i]=1
Neighbors=RegionQuery(i)
if len(Neighbors)>=MinPts:
C=C+1 #类别标志加一
ExpandCluster(i,Neighbors,C)
可视化图
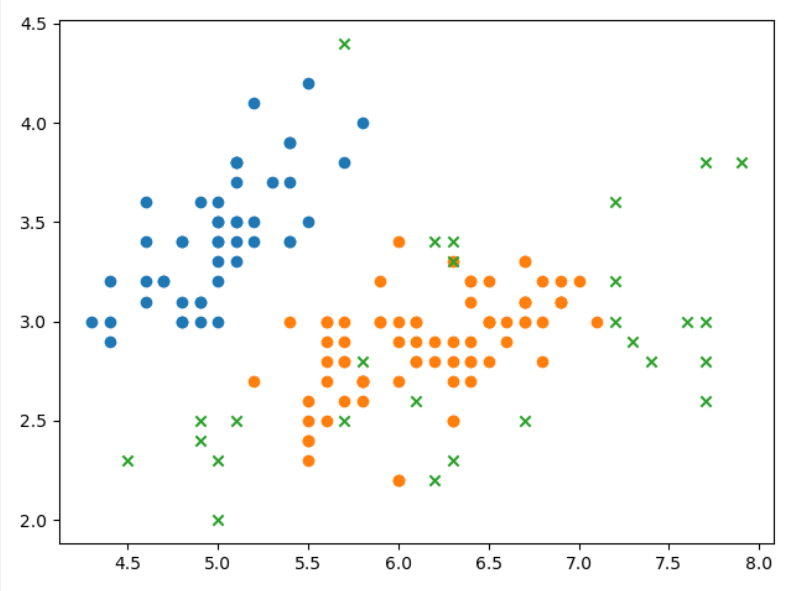
三种聚类算法结果的差异对比
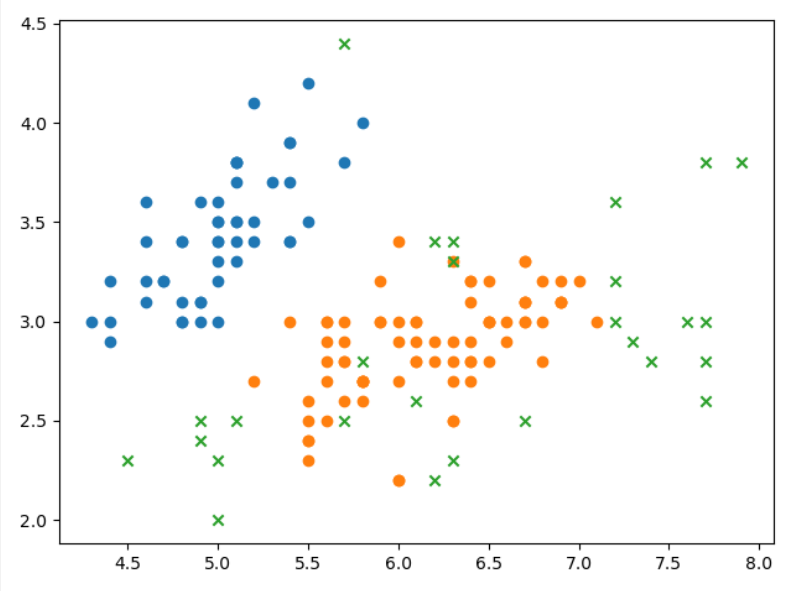
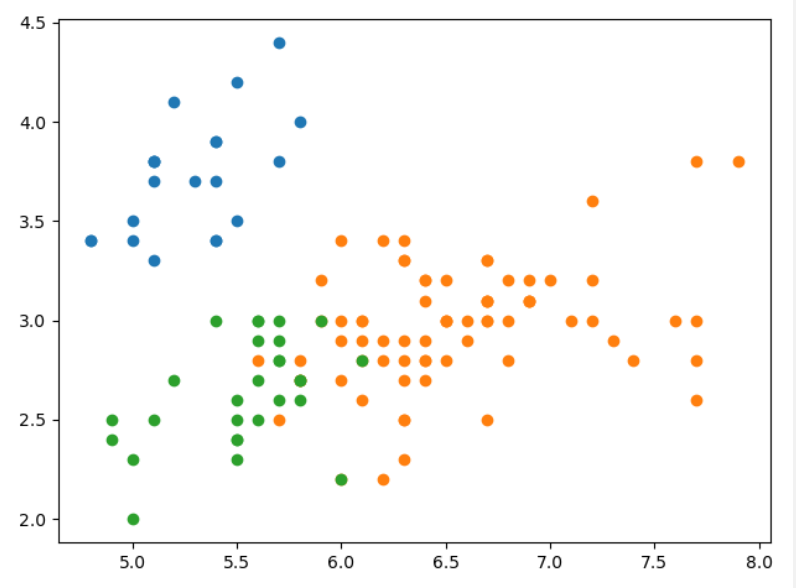
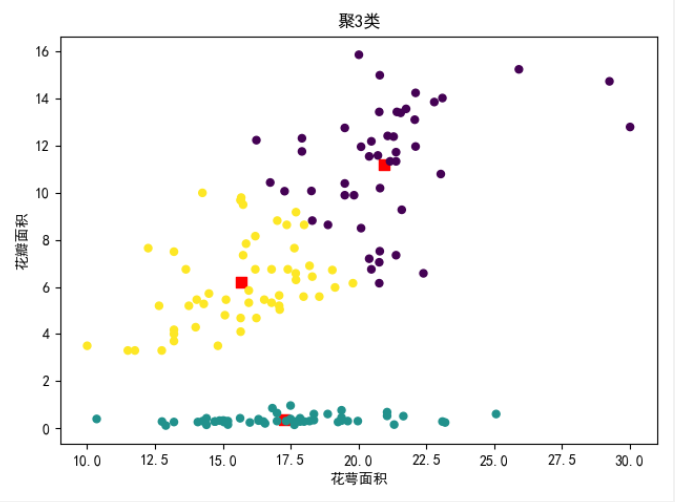
就目前的结果来看,$AGNES$ 的效果最好。当然,最后结果的呈现与参数的设置也有着极大的关系。
五、实验心得
通过这次实验,使我对 $K-means$,$AGNES$,$DBSCAN$ 等聚类算法有了一个更加深入的了解。并且在整个代码编写和参数调写的过程中,加强了我对算法的应用和认识,收获颇丰。
